Notícia
Pesquisa propõe novo método para o desenvolvimento de medicamentos contra câncer
Doutoranda da PUC-RS foi premiada internacionalmente por estudo que propõe a utilização da inteligência artificial para diminuir o tempo e o custo do desenvolvimento de novos fármacos
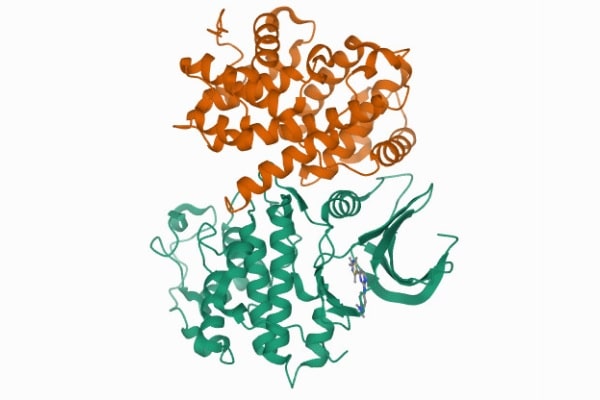
RCSB Protein Data Bank
Fonte
PUC-RS | Pontifícia Universidade Católica do Rio Grande do Sul
Data
sábado, 11 junho 2022 11:20
Áreas
Bioinformática. Biologia. Desenvolvimento de Fármacos. Indústria Farmacêutica. Inteligência Artificial. Microbiologia. Oncologia.
Docking molecular é um método computacional utilizado para pesquisar moléculas que possam se tornar medicamentos no futuro. Este método permite que sejam desenvolvidos estudos para entender como uma molécula se encaixa em determinada proteína e calcular a afinidade dessas duas. O docking, assim como todos os métodos computacionais vieram com a proposta de diminuir o tempo e o custo para desenvolver novos medicamentos, explicou Gabriela Bitencourt Ferreira, doutoranda do Programa de Pós-Graduação em Biologia Celular e Molecular da Pontifícia Universidade Católica do Rio Grande do Sul (PUC-RS).
De acordo com a doutoranda, normalmente leva em torno de 10 a 15 anos para desenvolver novos medicamentos, e o custo chega a 2,6 bilhões de dólares. Para isso, os métodos computacionais estão sendo utilizados para agilizar e otimizar este processo, pois poderiam indicar quais as melhores moléculas a serem testadas experimentalmente. Recentemente, Gabriela publicou um estudo (Exploring The Scoring Function Space For Structure-based Drug Design) como jovem pesquisadora no XXVIII Symposium on Bioinformatics and Computer-Aided Drug Discovery.
Sua pesquisa tem como foco o uso de inteligência artificial para o estudo de fármacos contra o câncer e foi premiada com a terceira colocação no evento que aconteceu em Moscou, capital da Rússia. De acordo com a aluna, os métodos computacionais ainda precisam de melhorias para que a informação gerada por eles seja mais confiável.
“Nosso trabalho buscou aprimorar os métodos computacionais usados atualmente para o desenvolvimento de medicamentos. A nossa intenção é que, depois de criarmos uma função escore específica para uma proteína, e comprovarmos que ela funciona, a gente possa usá-la para prever quais as moléculas possuem melhor afinidade com essa proteína e passar essa informação para os laboratórios experimentais. Dessa forma, ao invés dos laboratórios testarem mil moléculas eles poderiam testar apenas as 10 melhores que indicarmos”, explicou a pesquisadora.
Sobre a pesquisa
Gabriela explicou que o controle de cada programa de docking molecular é feito por uma equação chamada de função escore. De acordo com a doutoranda, as funções escores dos programas de docking são desenvolvidas pensando em diversas proteínas diferentes e são eficientes em determinar o encaixe de pequenas moléculas nas proteínas, mas que são insuficientes na hora de calcular a afinidade.
A doutoranda integra o Laboratório de Bioquímica Estrutural, coordenado pelo Dr. Walter Filgueira de Azevedo Junior, pesquisador da Escola de Ciências da Saúde e da Vida da PUC-RS. No laboratório foi criado o programa chamado SAnDReS, que possui mais de 60 métodos de aprendizado de máquina para fazer equações específicas para cada proteína. No Simpósio, Gabriela apresentou o projeto que mostra que esse método de desenvolver funções escores específicas para cada proteína melhora em até 10 vezes o cálculo de afinidade quando comparado com as funções escores usadas atualmente pelos programas de docking.
“Ou seja, a pesquisa atua com o foco de melhorar esse cálculo de afinidade. A nossa proposta é que, ao explorar o espaço de funções escore com métodos de inteligência artificial, a gente poderia ter uma equação adequada para cada proteína”, destacou a doutoranda.
Oportunidades de pesquisa desde a graduação
Gabriela é formada em Ciências Biológicas na PUCRS e atualmente integra o Programa de Pós-graduação em Biologia Celular e Molecular da Universidade. Durante sua graduação, a estudante participou do Programa de bolsas de Iniciação Científica e nesse programa teve oportunidade de trabalhar e aprender com o pesquisador Walter Filgueira. Gabriela ressaltou que aprendeu a apresentar trabalhos científicos e fazer resumos dos projetos nos Salões de Iniciação Científica e a PUCRS foi parte importante de todos os passos para que hoje ela conseguisse ser uma cientista.
Com sua participação estreante em um simpósio internacional, a doutoranda conquistou seu primeiro prêmio e incluiu novos métodos de validação em seu projeto graças ao contato com outros pesquisadores do mundo inteiro.
“Além do reconhecimento internacional que o meu projeto recebeu, eu também tive a oportunidade de discutir o meu projeto com cientistas renomados da área. Esse foi o meu primeiro simpósio e a minha primeira apresentação em inglês, então foi muito importante para poder ter essa oportunidade de conversar e explicar os meus resultados em outra língua também”, finalizou Gabriela.
Acesse a apresentação de Gabriela Ferreira no XXVIII Symposium on Bioinformatics and Computer-Aided Drug Discovery.
Acesse a notícia completa na página da PUC-RS.
Fonte: PUC-RS Pesquisa. Imagem: Proteína 2c5n. Fonte: RCSB Protein Data Bank.
Em suas publicações, o Canal Farma da Rede T4H tem o único objetivo de divulgação científica, tecnológica ou de informações comerciais para disseminar conhecimento. Nenhuma publicação do Canal Farma tem o objetivo de aconselhamento, diagnóstico, tratamento médico ou de substituição de qualquer profissional da área da saúde. Consulte sempre um profissional de saúde qualificado para a devida orientação, medicação ou tratamento, que seja compatível com suas necessidades específicas.
Os comentários constituem um espaço importante para a livre manifestação dos usuários, desde que cadastrados no Canal Farma e que respeitem os Termos e Condições de Uso. Portanto, cada comentário é de responsabilidade exclusiva do usuário que o assina, não representando a opinião do Canal Farma, que pode retirar, sem prévio aviso, comentários postados que não estejam de acordo com estas regras.





Por favor, faça Login para comentar