Notícia
Estudo usa Inteligência Artificial para acelerar a descoberta de novos fármacos
Resultados foram publicados na revista científica ‘Briefings In Bioinformatics’
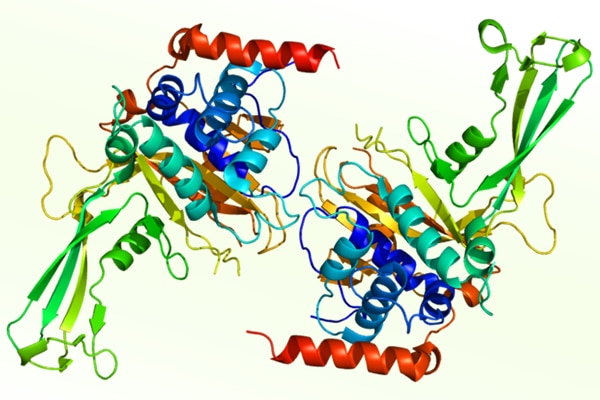
Emw via Wikimedia Commons
Fonte
Universidade de Coimbra
Data
quarta-feira, 14 setembro 2022 14:35
Áreas
Bioinformática. Biologia. Bioquímica. Desenvolvimento de Fármacos. Farmacologia. Indústria Farmacêutica. Microbiologia. Oncologia. Química Medicinal.
Uma equipe de pesquisadores da Universidade de Coimbra, em Portugal, criou um modelo computacional inovador que pode tornar mais rápido e menos dispendioso o desenvolvimento de novos fármacos para serem aplicados no tratamento de câncer, focados no contexto biológico da doença. Os resultados do estudo foram publicados na revista científica Briefings In Bioinformatics.
Considerando que a descoberta de um fármaco é um processo extremamente complexo, demorado e dispendioso, este trabalho, que resulta de uma colaboração entre a Faculdade de Ciências e Tecnologia (FCTUC) e a Faculdade de Farmácia (FFUC) da Universidade de Coimbra, teve como objetivo encurtar as etapas iniciais de desenvolvimento de fármacos, recorrendo à Inteligência Artificial (IA), através de métodos computacionais que consigam gerar compostos farmacologicamente interessantes de uma forma mais rápida e automatizada.
Para desenvolver o novo modelo, a equipe do Departamento de Engenharia Informática da FCTUC recorreu a técnicas de Machine Learning, notadamente Deep Learning – um método que utiliza redes neuronais artificiais. Estas estruturas permitem criar modelos inteligentes “através da mimetização da capacidade de aprendizagem dos modelos biológicos. Deste modo são capazes de identificar padrões embebidos em conjuntos de dados e, a partir daí, é possível obter modelos que geram novas estruturas moleculares e que preveem propriedades biológicas de interesse”, explicou Tiago Oliveira Pereira, primeiro autor do estudo que faz parte do seu doutoramento, orientado pela pesquisadora Dra. Maryam Abbasi e pelo Dr. Joel P. Arrais.
Os pesquisadores utilizaram também o chamado Reinforcement Learning (aprendizagem por reforço), que permite otimizar o modelo de geração durante a exploração do espaço químico existente. “À medida que o modelo gera novas moléculas, ele recebe uma recompensa, que será maior ou menor, dependendo do estado de otimização das propriedades dos compostos. Assim, ao longo deste processo de otimização, o gerador de compostos vai aprender a identificar as regiões do espaço químico que lhe permitam obter maior recompensa e melhores compostos”, explicou o pesquisador da FCTUC.
O modelo desenvolvido é inovador porque, explicaram os autores, “é um modelo que combina informação química, através dos compostos, e biológica, por via de informação da expressão gênica, de modo a encontrar moléculas promissoras na inibição do receptor e que não causem efeitos indesejados ao sistema biológico”.
Com a colaboração do laboratório do professor Dr. Jorge Salvador da FFUC, foi possível aplicar o modelo em um caso de estudo para a geração de compostos capazes de inibir a proteína USP7 (Ubiquitin specific protease 7). Esta proteína, sublinhou Tiago Oliveira Pereira, assume um papel fundamental “na progressão de vários tipos de câncer e, atualmente, é vista como um receptor importante para o desenvolvimento de fármacos”.
Os resultados obtidos nas experiências realizadas são altamente promissores, tendo o modelo demonstrado elevada capacidade para gerar moléculas potenciais inibidoras da USP7. “Mais de 90% das moléculas continham propriedades físicas, químicas e biológicas essenciais para que ocorra a interação com o receptor. Para além disto, verificamos que alguns compostos gerados pelo modelo apresentam semelhanças com fármacos anticâncer ao nível dos seus grupos ativos, o que valida a abordagem implementada”, destacou Tiago Pereira.
Apesar de ter sido validado com dados de câncer de mama, o novo modelo computacional pode ser aplicado a “diversos contextos em que se possam obter dados de expressão gênica associados à progressão da doença”, explicou o pesquisador, adiantando ainda que os próximos passos do estudo vão incidir na melhoria da arquitetura implementada e na “definição de um conjunto de métodos de validação para filtrar as moléculas obtidas e, dependendo dos resultados, avançar para a síntese dos melhores compostos”.
Acesse o resumo do artigo científico (em inglês).
Acesse a notícia completa na página da Universidade de Coimbra.
Fonte: Cristina Pinto, FCTUC/Universidade de Coimbra. Imagem: Estrutura da proteína USP7. Fonte: Emw via Wikimedia Commons.
Em suas publicações, o Canal Farma da Rede T4H tem o único objetivo de divulgação científica, tecnológica ou de informações comerciais para disseminar conhecimento. Nenhuma publicação do Canal Farma tem o objetivo de aconselhamento, diagnóstico, tratamento médico ou de substituição de qualquer profissional da área da saúde. Consulte sempre um profissional de saúde qualificado para a devida orientação, medicação ou tratamento, que seja compatível com suas necessidades específicas.
Os comentários constituem um espaço importante para a livre manifestação dos usuários, desde que cadastrados no Canal Farma e que respeitem os Termos e Condições de Uso. Portanto, cada comentário é de responsabilidade exclusiva do usuário que o assina, não representando a opinião do Canal Farma, que pode retirar, sem prévio aviso, comentários postados que não estejam de acordo com estas regras.





Por favor, faça Login para comentar