Notícia
Pesquisadores desenvolvem nova ferramenta para geração e design de sequências de proteínas
Nova técnica usa modelo de linguagem de proteína para gerar sequências de proteínas com propriedades comparáveis às sequências naturais
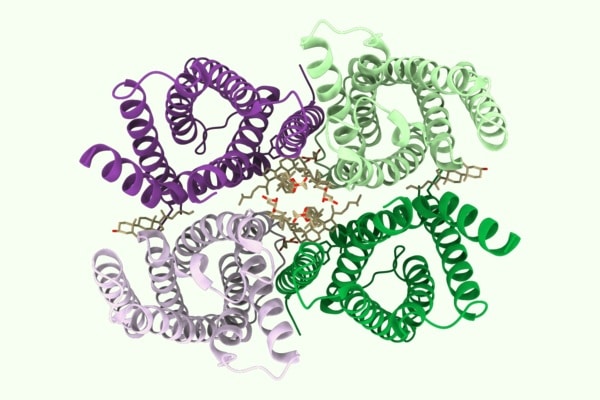
Batuujin Burendei via Wikimedia Commons
Fonte
EPFL | Escola Politécnica Federal de Lausanne
Data
terça-feira, 14 março 2023 16:10
Áreas
Bioinformática. Biologia. Bioquímica. Inteligência Artificial. Microbiologia. Proteômica.
Projetar novas proteínas com estrutura e função específicas é um objetivo altamente importante da bioengenharia, mas o vasto tamanho do espaço da sequência de proteínas dificulta a busca por novas proteínas. No entanto, um novo estudo do grupo de pesquisa da Dra. Anne-Florence Bitbol, pesquisadora na Escola de Ciências da Vida da Escola Politécnica Federal de Lausanne (EPFL), na Suíça, descobriu que uma rede neural de aprendizado profundo, o modelo MSA Transformer, pode ser uma solução promissora.
Desenvolvido em 2021, o modelo MSA Transformer funciona de forma semelhante ao processamento de linguagem natural, utilizado, por exemplo, no ChatGPT. A equipe, composta pelo doutorando Damiano Sgarbossa e Dr. Umberto Lupo, além da própria Dra. Anne-Florence Bitbol, propôs e testou um ‘método iterativo’, que se baseia na capacidade do modelo de prever partes ausentes ou mascaradas de uma sequência com base no contexto circundante.
A equipe descobriu que, por meio dessa abordagem, o MSA Transformer pode ser usado para gerar novas sequências de proteínas a partir de determinadas ‘famílias’ de proteínas (grupos de proteínas com sequências semelhantes), com propriedades semelhantes às sequências naturais.
De fato, as sequências de proteínas geradas a partir de grandes famílias com muitos homólogos tinham propriedades melhores ou semelhantes às sequências geradas pelos modelos de Potts. “Um modelo de Potts é um tipo totalmente diferente de modelo generativo não baseado em processamento de linguagem natural ou aprendizado profundo, que foi recentemente validado experimentalmente”, explicou a professora Anne Bitbol. “Nossa nova abordagem baseada no MSA Transformer nos permitiu gerar proteínas até mesmo de famílias pequenas, onde os modelos de Potts funcionam mal.”
O modelo MSA Transformer reproduz as estatísticas de ordem superior e a distribuição de sequências em dados naturais com mais precisão do que outros modelos, o que o torna um forte candidato para geração de sequência de proteínas e design de proteínas.
“Esse trabalho pode levar ao desenvolvimento de novas proteínas com estruturas e funções específicas; esperamos que tais abordagens permitam aplicações médicas importantes no futuro”, disse a Dra. Anne Bitbol. “O potencial do MSA Transformer como um forte candidato para o design de proteínas oferece novas possibilidades empolgantes para o campo da bioengenharia”.
O estudo foi publicado na revista científica eLife, cujos editores comentaram: “Este importante estudo propõe um método para amostrar novas sequências [a partir] de um modelo de linguagem de proteínas que pode ter aplicações interessantes no design de sequências de proteínas. As reivindicações são apoiadas por um sólido benchmarking das sequências projetadas em termos de qualidade, novidade e diversidade”.
Acesse o artigo científico completo (em inglês).
Acesse a notícia completa na página da Escola Politécnica Federal de Lausanne (em inglês).
Fonte: Nik Papageorgiou, EPFL. Imagem: PDB 6nf4. Fonte: Batuujin Burendei via Wikimedia Commons.
Em suas publicações, o Canal Farma da Rede T4H tem o único objetivo de divulgação científica, tecnológica ou de informações comerciais para disseminar conhecimento. Nenhuma publicação do Canal Farma tem o objetivo de aconselhamento, diagnóstico, tratamento médico ou de substituição de qualquer profissional da área da saúde. Consulte sempre um profissional de saúde qualificado para a devida orientação, medicação ou tratamento, que seja compatível com suas necessidades específicas.
Os comentários constituem um espaço importante para a livre manifestação dos usuários, desde que cadastrados no Canal Farma e que respeitem os Termos e Condições de Uso. Portanto, cada comentário é de responsabilidade exclusiva do usuário que o assina, não representando a opinião do Canal Farma, que pode retirar, sem prévio aviso, comentários postados que não estejam de acordo com estas regras.





Por favor, faça Login para comentar