Notícia
Infectar micróbios intestinais com vírus carregado com CRISPR demonstra potencial para edição de genes do microbioma
Pesquisadores foram capazes de remover ‘pedaços’ de genes da bactéria Escherichia coli de camundongos e mudar a composição geral das comunidades bacterianas que povoam seus sistemas digestivos
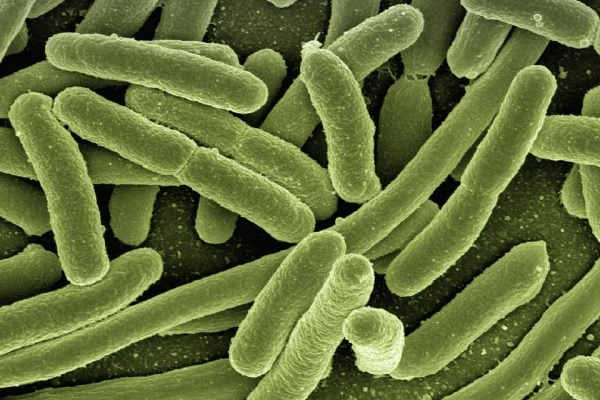
Pixabay
Fonte
Universidade da Califórnia em San Francisco
Data
quinta-feira, 2 dezembro 2021 11:15
Áreas
Biologia. Genoma. Imunologia. Microbiologia.
Pesquisadores da Universidade da Califórnia em San Francisco (USCF), nos Estados Unidos, usaram com sucesso o sistema CRISPR de edição de DNA para alterar os genomas de bactérias que vivem no intestino de mamíferos, um desenvolvimento que pode avançar a compreensão do microbioma e pode, eventualmente, abrir caminho para tratamentos de doenças relacionadas ao intestino.
No estudo, publicado na revista científica Cell Reports, os pesquisadores foram capazes de remover pedaços de genes da bactéria Escherichia coli de camundongos e mudar a composição geral das comunidades bacterianas que povoam seus sistemas digestivos.
“Demonstramos a primeira edição de gene estável no microbioma intestinal de um mamífero”, disse o Dr. Peter Turnbaugh, professor de Microbiologia e Imunologia da UCSF. “Este é o ponto de partida para tentar criar bactérias dentro do intestino.”
Atualmente, pesquisadores e médicos que desejam alterar o micróbio intestinal têm opções extremamente limitadas. A intoxicação alimentar bacteriana e problemas semelhantes podem ser tratados com antibióticos de amplo espectro, por exemplo, mas esse medicamento acaba matando muitos micróbios “bons” junto com os maus. Os transplantes fecais também têm sido usados na esperança de ressemear um microbioma saudável em pacientes com certas infecções e doenças gastrointestinais. Mas os médicos não podem ter certeza de que os micróbios introduzidos conseguirão superar as comunidades bacterianas existentes do paciente, o que significa que o tratamento nem sempre é bem-sucedido.
Modificar bactérias que já prosperam no sistema digestivo será, portanto, uma abordagem chave na pesquisa e no tratamento de problemas de saúde relacionados ao microbioma no futuro, disse o Dr. Turnbaugh. Alterar diretamente os genomas de micróbios no intestino introduziria um nível de precisão nos tratamentos de microbioma que ainda não foi possível.
“Ser capaz de alterar o DNA de micróbios que já estão no intestino pode nos permitir estudar o microbioma de uma forma mais controlada do que antes”, ressaltou o pesquisador.
O estudo do Dr. Turnbaugh se concentrou na E. coli, uma bactéria que ocorre naturalmente no intestino, mas que mantém uma má reputação porque certas cepas podem causar intoxicação alimentar. Uma aplicação útil da edição de genes de precisão no microbioma intestinal seria visar cepas prejudiciais de E. coli, deixando as úteis intactas.
Para este estudo, os pesquisadores queriam saber se eles poderiam usar a ferramenta de edição de genes para direcionar e matar uma cepa de E. coli enquanto deixava outra isolada. A equipe do Dr. Turnbaugh usou um vírus, chamado M13, para injetar um sistema CRISPR-Cas9 nas células de uma cepa específica de E. coli, onde começou a trabalhar cortando segmentos de DNA.
Os resultados foram dramáticos. Antes do sistema CRISPR-Cas9 ser introduzido, a cepa alvo era mais proeminente nas amostras fecais coletadas dos camundongos no experimento. Após a edição do gene, no entanto, a cepa-alvo começou a desaparecer rapidamente. Após duas semanas, representou apenas 1% da população de células monitoradas.
Uma chave para o sucesso do estudo foi o uso de uma forma transformada por engenharia do M13, um vírus que ataca naturalmente a E. coli, mas normalmente não sobrevive bem no sistema digestivo. Para resolver esse problema, o Dr. Turnbaugh e seus colegas aplicaram um antibiótico com gene de resistência no DNA que o M13 entregaria às células infectadas, permitindo que o vírus – e o sistema CRISPR-Cas9 que ele carregava – se propagasse mais facilmente.
O pesquisador prevê que uma abordagem semelhante poderia algum dia ser usada para promover o crescimento de bactérias intestinais “boas” em humanos. Por exemplo, se os pesquisadores editassem genes em certas cepas de bactérias para permitir que as bactérias se alimentassem de nutrientes raros, uma pessoa poderia ganhar algum controle sobre a mistura de micróbios que florescem em seu intestino simplesmente adicionando quantidades desses nutrientes à sua dieta. Primeiro, disse ele, os pesquisadores precisarão expandir a lista de vírus em seu kit de ferramentas e experimentar como a alteração de membros individuais do microbioma afeta a população de bactérias em geral.
“O sonho é que você poderia simplesmente escolher quais cepas específicas em seu intestino – ou mesmo apenas genes individuais – você deseja promover ou eliminar. Estamos muito entusiasmados com o quão longe fomos capazes de fazer isso com a E. coli. Esperançosamente, isso levará a ferramentas semelhantes para outros membros da microbiota intestinal”, concluiu o Dr. Peter Turnbaugh.
Acesse o artigo científico completo (em inglês).
Acesse a notícia completa na página da UCSF (em inglês).
Fonte: Lindzi Wessel, UCSF. Imagem: Bactéria Escherichia coli (E. Coli). Fonte: Pixabay.
Em suas publicações, o Canal Farma da Rede T4H tem o único objetivo de divulgação científica, tecnológica ou de informações comerciais para disseminar conhecimento. Nenhuma publicação do Canal Farma tem o objetivo de aconselhamento, diagnóstico, tratamento médico ou de substituição de qualquer profissional da área da saúde. Consulte sempre um profissional de saúde qualificado para a devida orientação, medicação ou tratamento, que seja compatível com suas necessidades específicas.
Os comentários constituem um espaço importante para a livre manifestação dos usuários, desde que cadastrados no Canal Farma e que respeitem os Termos e Condições de Uso. Portanto, cada comentário é de responsabilidade exclusiva do usuário que o assina, não representando a opinião do Canal Farma, que pode retirar, sem prévio aviso, comentários postados que não estejam de acordo com estas regras.





Por favor, faça Login para comentar