Notícia
Tipos de células ‘invisíveis’ e expressão genética são reveladas com melhoria na análise de dados de sequenciamento
Identificar diferentes tipos de células é fundamental para compreender o vasto número de funções desempenhadas pelo corpo, desde processos saudáveis, como a sensação de sede, até o mau funcionamento celular em doenças
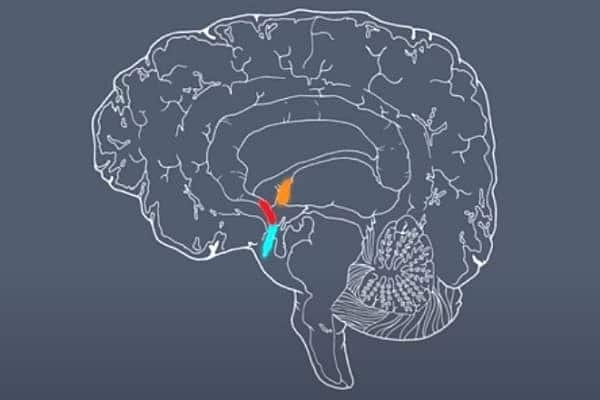
Reprodução, Caltech
Fonte
Caltech | Instituto de Tecnologia da Califórnia
Data
quarta-feira, 13 setembro 2023 17:40
Áreas
Bioinformática. Biologia. Biotecnologia. Computação. Engenharia Biológica. Genética. Genômica. Microbiologia.
Em 2018, pesquisadores do laboratório do Dr. Yuki Oka, professor de Biologia e pesquisador do Heritage Medical Research Institute do Instituto de Tecnologia da Califórnia (Caltech), nos Estados Unidos, fizeram uma grande descoberta: identificaram um tipo de neurônio envolvido na saciedade da sede. Mas eles estavam enfrentando um problema: uma técnica de última geração chamada sequenciamento de RNA unicelular (scRNA-seq) não conseguia encontrar esses neurônios relacionados à sede em amostras de tecido cerebral (especificamente, de uma região chamada núcleo pré-óptico medial), que deveria contê-los.
“Sabíamos que a marcação genética que adicionamos aos nossos neurônios caracterizados estava sendo expressa no núcleo pré-óptico medial do cérebro, mas não vimos o gene quando traçamos o perfil daquela região do cérebro com scRNA-seq. Ouvimos isso de muitos colegas – faltavam tipos de células e expressões genéticas no scRNA-seq que eles sabiam que deveriam estar lá. E começamos a nos perguntar por que isso acontecia”, disse o professor Yuki Oka.
Identificar diferentes tipos de células é fundamental para compreender o vasto número de funções desempenhadas pelo corpo, desde processos saudáveis, como a sensação de sede, até o mau funcionamento celular em doenças. Por exemplo, muitos pesquisadores estão atualmente à procura de tipos de células que possam estar ligados a doenças específicas, como a doença de Parkinson. Determinar os tipos precisos de células envolvidas em tais processos é fundamental para todos esses estudos. Agora, uma colaboração entre o laboratório do professor Oka do Caltech e o laboratório do Dr. Allan-Hermann Pool, pesquisador do Southwestern Medical Center da Universidade do Texas, também nos Estados Unidos, demonstrou como otimizar uma etapa fundamental na análise de scRNA-seq para recuperar tipos de células ausentes e dados de expressão gênica que geralmente são descartados.
Um artigo descrevendo o trabalho foi publicado na revista científica Nature Methods.
“Melhoramos a análise dos dados de sequenciamento de RNA unicelular de última geração, revelando a expressão de centenas ou até milhares de genes para conjuntos de dados individuais”, disse o professor Yuki Oka. “É importante permitir este tipo de precisão porque os processos biológicos são ricos e complicados. Pesquisas recentes identificaram mais de 5.000 tipos distintos de neurônios no cérebro de um camundongo, e o cérebro humano é presumivelmente mais complexo. Precisamos que nossas técnicas sejam muito sensíveis e o mais abrangentes possível.”
Compreendendo a expressão genética
Existem trilhões de células no corpo, cada uma desempenhando as diversas funções que permitem a vida – ou, em alguns casos, que levam à doença. As células são diferenciadas umas das outras por sua função. Por exemplo, as células T assassinas do sistema imunitário procuram e destroem os agentes patogênicos que causam doenças, os neurônios disparam sinais elétricos que estão na base da função cerebral e as células da pele agrupam-se firmemente para criar uma barreira contra o mundo exterior. Os pesquisadores identificaram atualmente milhares de tipos de células distintos, mas outras variedades únicas provavelmente permanecem desconhecidas.
Embora as células possam diferir em forma e função, a maioria das células de um determinado organismo contém um modelo genético idêntico – o genoma. O genoma contém instruções sobre como realizar qualquer tarefa celular. Os genes que compõem o genoma estão escritos no DNA, localizado no núcleo da célula. Os genes expressos são copiados no RNA, que é transportado para fora do núcleo e para o resto da célula para realizar funções.
Em qualquer célula (e tipo de célula), apenas um determinado subconjunto de genes é expresso, ou ativado, num determinado momento. Essas variações na expressão gênica resultam em diferenças nos tipos de células.
Melhorando técnicas para estimativa da expressão gênica
A scRNA-seq é uma técnica poderosa para identificar tipos de células. Com esse método, uma célula é aberta e a informação genética expressa em seu interior é rotulada com uma etiqueta molecular que serve como um ‘código de barras’. O scRNA-seq pode fazer isso rapidamente para milhares de células em uma única amostra de tecido, com cada célula recebendo seu próprio código de barras exclusivo. A análise computacional pode então ser realizada para determinar quais conjuntos de genes são expressos em células individuais, e modelos de computador podem avaliar esses dados para procurar padrões e identificar tipos de células distintos.
Um problema com a técnica, no entanto, era que certos RNAs normalmente não eram incluídos nas estimativas de expressão genética, embora representassem genes expressos.
O motivo, como descobriram os pesquisadores, está relacionado a um problema com o chamado ‘transcriptoma de referência’, para o qual os pesquisadores mapeiam os dados de sequenciamento. Por exemplo, os investigadores estudaram extensivamente o genoma de camundongos e rotularam-no ou anotaram-no detalhadamente, criando uma referência digital, ou ‘transcriptoma’, que mapeia sequências de DNA e os seus genes correspondentes.
Esta anotação, descobriram os pesquisadores, deve ser otimizada para scRNA-seq para evitar a perda de informações de expressão gênica – que pode surgir se os genes localizados nas extremidades de uma cadeia de DNA forem mal anotados, por exemplo, ou se houver extensa sobreposição entre transcrições de genes vizinhos. Tais complicações podem impedir a detecção de milhares de genes. (Estas questões são particularmente pronunciadas quando se utilizam formas de scRNA-seq de alto rendimento que, para reduzir custos, examinam apenas a extremidade final dos genes; a maioria dos atlas que foram criados para descrever a complexidade celular dos nossos tecidos baseiam-se nestes métodos.)
Precisão e alta resolução são extremamente importantes na identificação de tipos de células distintos. Por exemplo, digamos que duas células expressem cada uma os genes ‘A’, ‘B’, ‘C’ e ‘D’, mas apenas uma célula expressa o gene ‘E’ enquanto a outra não. Se uma técnica de sequenciamento não capturar a expressão de ‘E’, então os dados sugeririam que as duas células são idênticas, quando na verdade não são.
Liderada pelo Dr. Allan Pool, ex-bolsista de pós-doutorado do Caltech e primeiro autor do estudo, a equipe otimizou o transcriptoma de referência para os genomas de camundongos e humanos e, ao longo de vários anos, construiu uma estrutura computacional para fixar os transcriptomas de referência de outros organismos.
Acesse o resumo do artigo científico (em inglês).
Acesse a notícia completa na página do Instituto de Tecnologia da Califórnia (em inglês).
Fonte: Lori Dajose, Caltech. Imagem: três regiões do cérebro que processam a sede. Fonte: Reprodução, Caltech.
Em suas publicações, o Canal Farma da Rede T4H tem o único objetivo de divulgação científica, tecnológica ou de informações comerciais para disseminar conhecimento. Nenhuma publicação do Canal Farma tem o objetivo de aconselhamento, diagnóstico, tratamento médico ou de substituição de qualquer profissional da área da saúde. Consulte sempre um profissional de saúde qualificado para a devida orientação, medicação ou tratamento, que seja compatível com suas necessidades específicas.
Os comentários constituem um espaço importante para a livre manifestação dos usuários, desde que cadastrados no Canal Farma e que respeitem os Termos e Condições de Uso. Portanto, cada comentário é de responsabilidade exclusiva do usuário que o assina, não representando a opinião do Canal Farma, que pode retirar, sem prévio aviso, comentários postados que não estejam de acordo com estas regras.





Por favor, faça Login para comentar