Notícia
Aprendizado de máquina pode identificar cânceres não diagnosticáveis
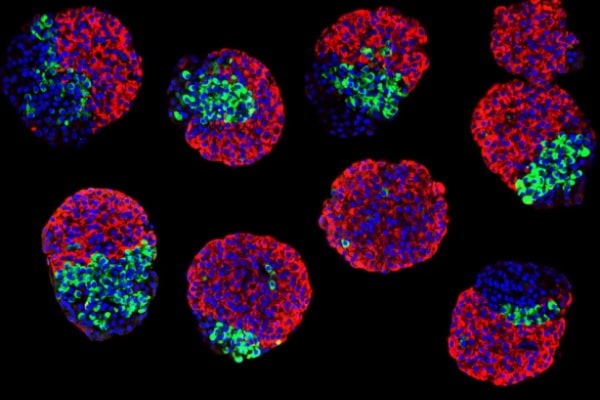
Novo modelo que mapeia caminhos de desenvolvimento para células tumorais pode desvendar identidade de cânceres de origem desconhecida
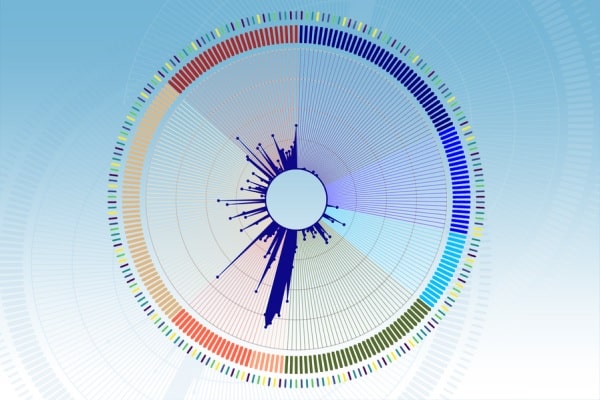
Divulgação, MIT
Fonte
MIT | Instituto de Tecnologia de Massachusetts
Data
quarta-feira, 7 setembro 2022 12:10
Áreas
Bioinformática. Biologia. Biomedicina. Biotecnologia. Computação. Diagnóstico. Genoma. Inteligência Artificial. Medicina Personalizada. Modelagem Matemática. Oncologia. Radiologia. Saúde Pública.
O primeiro passo na escolha do tratamento adequado para um paciente com câncer é identificar o tipo específico de câncer, incluindo a determinação do local primário – o órgão ou parte do corpo onde o câncer começou.
Em casos raros, a origem de um câncer não pode ser determinada, mesmo com testes extensivos. Embora esses cânceres de origem desconhecida tendam a ser agressivos, os oncologistas devem tratá-los com terapias não direcionadas, que frequentemente apresentam toxicidade severa e resultam em baixas taxas de sobrevida.
Uma nova abordagem de aprendizado profundo desenvolvida por pesquisadores do Instituto Koch para Pesquisa Integrativa do Câncer no Instituto de Tecnologia de Massachusetts (MIT) e do Massachusetts General Hospital (MGH) pode ajudar a classificar cânceres primários desconhecidos, analisando mais de perto a expressão gênica relacionada ao desenvolvimento e diferenciação celular precoce.
“Às vezes, você pode aplicar todas as ferramentas que os patologistas têm a oferecer e ainda fica sem uma resposta”, disse o Dr. Salil Garg, pesquisador clínico do Instituto Koch e patologista do MGH. “Ferramentas de aprendizado de máquina como esta podem capacitar os oncologistas a escolher tratamentos mais eficazes e dar mais orientação aos seus pacientes.”
O Dr. Salil Garg é o autor sênior de um novo estudo, publicado recentemente na revista científica Cancer Discovery. O Dr. Enrico Moiso, pós-doutorando do MIT, é o autor principal do artigo. A ferramenta de inteligência artificial é capaz de identificar tipos de câncer com alto grau de sensibilidade e precisão.
Aprendizado de máquina em desenvolvimento
Analisar as diferenças na expressão gênica entre os diferentes tipos de tumores primários desconhecidos é um problema ideal para o aprendizado de máquina resolver. As células cancerosas parecem e se comportam de maneira bastante diferente das células normais, em parte por causa de extensas alterações na forma como seus genes são expressos. Graças aos avanços no perfil de uma única célula e aos esforços para catalogar diferentes padrões de expressão celular em atlas de células, existem dados disponíveis que contêm pistas de como e de onde os diferentes tipos de câncer se originaram.
No entanto, construir um modelo de aprendizado de máquina que aproveite as diferenças entre células saudáveis e normais e entre diferentes tipos de câncer em uma ferramenta de diagnóstico é um ato de equilíbrio. Se um modelo é muito complexo e leva em conta muitas características da expressão do gene do câncer, o modelo pode parecer aprender os dados de treinamento perfeitamente, mas errar quando encontra novos dados. No entanto, ao simplificar o modelo e restringir o número de recursos, o modelo pode perder os tipos de informações que levariam a classificações precisas dos tipos de câncer.
A fim de encontrar um equilíbrio entre reduzir o número de recursos e ainda extrair as informações mais relevantes, a equipe concentrou o modelo em sinais de vias de desenvolvimento alteradas em células cancerígenas. À medida que um embrião se desenvolve e as células indiferenciadas se especializam em vários órgãos, uma infinidade de caminhos direciona como as células se dividem, crescem, mudam de forma e migram. À medida que o tumor se desenvolve, as células cancerosas perdem muitas das características especializadas de uma célula madura. Ao mesmo tempo, elas começam a se assemelhar às células embrionárias de algumas maneiras, pois ganham a capacidade de proliferar, transformar e metastatizar para novos tecidos. Muitos dos programas de expressão gênica que impulsionam a embriogênese são conhecidos por serem reativados ou desregulados em células cancerígenas.
Os pesquisadores compararam dois atlas de células grandes, identificando correlações entre células tumorais e embrionárias: o Cancer Genome Atlas (TCGA), que contém dados de expressão gênica para 33 tipos de tumores, e o Mouse Organogenesis Cell Atlas (MOCA), que traça o perfil de 56 trajetórias separadas de células embrionárias à medida que se desenvolvem e se diferenciam.
“As ferramentas de resolução de célula única mudaram drasticamente a forma como estudamos a biologia do câncer, mas como tornamos essa revolução impactante para os pacientes é outra questão”, explicou o Dr. Enrico Moiso. “Com o surgimento de atlas de células de desenvolvimento, especialmente aqueles que se concentram nas fases iniciais da organogênese, como o MOCA, podemos expandir nossas ferramentas além das informações histológicas e genômicas e abrir portas para novas formas de perfilar e identificar tumores e desenvolver novos tratamentos.”
O mapa resultante de correlações entre padrões de expressão de genes de desenvolvimento em células tumorais e embrionárias foi então transformado em um modelo de aprendizado de máquina. Os pesquisadores dividiram a expressão gênica de amostras de tumor do TCGA em componentes individuais que correspondem a um ponto específico do tempo em uma trajetória de desenvolvimento e atribuíram a cada um desses componentes um valor matemático. Os pesquisadores então construíram um modelo de aprendizado de máquina, chamado Developmental Multilayer Perceptron (D-MLP), que classifica um tumor por seus componentes de desenvolvimento e, em seguida, prevê sua origem.
Embora o estudo apresente uma abordagem poderosa para classificar tumores, ele tem algumas limitações. Em trabalhos futuros, os pesquisadores planejam aumentar o poder preditivo do modelo incorporando outros tipos de dados, principalmente informações obtidas de radiologia, microscopia e outros tipos de imagens de tumores.
“A expressão gênica de desenvolvimento representa apenas uma pequena fatia de todos os fatores que podem ser usados para diagnosticar e tratar cânceres. Integrar informações de radiologia, patologia e expressão genética é o verdadeiro próximo passo na medicina personalizada para pacientes com câncer”, concluiu o Dr. Salil Garg.
Acesse o artigo científico completo (em inglês).
Acesse a notícia completa na página do Instituto de Tecnologia de Massachusetts (em inglês).
Fonte: Koch Institute/MIT. Imagem: Divulgação, MIT.
Em suas publicações, o Canal Farma da Rede T4H tem o único objetivo de divulgação científica, tecnológica ou de informações comerciais para disseminar conhecimento. Nenhuma publicação do Canal Farma tem o objetivo de aconselhamento, diagnóstico, tratamento médico ou de substituição de qualquer profissional da área da saúde. Consulte sempre um profissional de saúde qualificado para a devida orientação, medicação ou tratamento, que seja compatível com suas necessidades específicas.
Os comentários constituem um espaço importante para a livre manifestação dos usuários, desde que cadastrados no Canal Farma e que respeitem os Termos e Condições de Uso. Portanto, cada comentário é de responsabilidade exclusiva do usuário que o assina, não representando a opinião do Canal Farma, que pode retirar, sem prévio aviso, comentários postados que não estejam de acordo com estas regras.


Por favor, faça Login para comentar