Notícia
Cientistas usam Inteligência Artificial em engenharia de interações moleculares
Aplicando Inteligência Artificial para caracterizar milhões de fragmentos de proteínas, pesquisadores projetaram computacionalmente novos ligantes de proteínas para alvos-chave, incluindo a proteína spike do vírus SARS-CoV-2

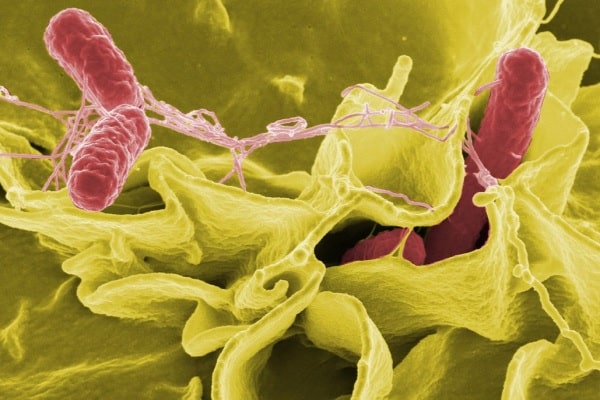
EPFL/Ella Maru Studio
Fonte
EPFL | Escola Politécnica Federal de Lausanne
Data
sexta-feira, 5 maio 2023 06:25
Áreas
Bioinformática. Biologia. Bioquímica. Biotecnologia. Computação. Imunoterapia. Inteligência Artificial. Medicina de Precisão. Microbiologia. Proteômica. Saúde Pública.
Em 2019, cientistas do Laboratório de Design de Proteínas e Imunoengenharia (LPDI) da Escola de Engenharia e da Escola de Ciências da Vida da Escola Politécnica Federal de Lausanne (EPFL), na Suíça, liderados pelo Dr. Bruno Correia, desenvolveram o MaSIF: um método baseado em aprendizado de máquina para escanear milhões de superfícies de proteínas em minutos para analisar sua estrutura e propriedades funcionais. O objetivo final dos pesquisadores era projetar computacionalmente as interações de proteínas, encontrando correspondências ideais entre as moléculas com base em suas ‘impressões digitais’ químicas e geométricas de superfície.
Quatro anos depois, eles conseguiram exatamente isso. Em um artigo publicado na revista científica Nature, eles relataram a criação de novas proteínas chamadas ligantes, projetadas para interagir com quatro alvos de proteínas terapeuticamente relevantes, incluindo a proteína spike do SARS-CoV-2.
Projetando uma correspondência molecular perfeita
As interações físicas entre as proteínas influenciam qualquer coisa, desde a sinalização celular e o crescimento até as respostas imunes, portanto, a capacidade de controlar as interações proteína-proteína é de grande interesse para os campos da biologia e da biotecnologia. Embora as representações dos livros didáticos sobre a ligação de proteínas possam parecer tão simples quanto encaixar peças de quebra-cabeça, a realidade é mais complexa: as superfícies das proteínas variam amplamente e são dinâmicas, tornando difícil prever como e onde os eventos de ligação ocorrerão.
“Uma peça de quebra-cabeça é bidimensional, mas com superfícies de proteínas, estamos olhando para múltiplas dimensões: composição química, como interações de carga positiva versus negativa; complementaridade de formas, curvatura, etc.”, explicou Anthony Marchand, doutorando do LPDI e coautor do estudo.
Para projetar novos ligantes de proteínas, os pesquisadores usaram o MaSIF para criar ‘impressões digitais’ da superfície da proteína e, em seguida, identificaram superfícies complementares para os principais locais-alvo de proteínas a partir de um banco de dados de fragmentos. Eles então enxertaram digitalmente os fragmentos em andaimes de proteína maiores e selecionaram os ligantes resultantes previstos para interagir melhor com seus alvos. Depois de sintetizar e testar esses ligantes selecionados no laboratório, os pesquisadores puderam confirmar a hipótese gerada computacionalmente.
“O fato de sermos capazes de projetar novos ligantes de proteínas específicos do local em apenas alguns meses torna esse método muito interessante para a terapêutica. Não é apenas uma ferramenta: é um pipeline”, destacou Anthony Marchand.
‘Direto do computador’
Os pesquisadores estavam desenvolvendo ligantes de proteína para três principais alvos de imunoterapia contra o câncer quando a pandemia de COVID-19 começou, então eles adicionaram a proteína spike SARS-CoV-2 à sua lista. Usando sua abordagem, os quatro ligantes que eles produziram mostraram excelentes afinidades com seus alvos.
A taxa de sucesso do MaSIF, combinada com sua velocidade e capacidade de produzir designs específicos de local de alta qualidade, demonstram seu potencial terapêutico. Por exemplo, a capacidade de gerar ligantes de proteínas precisos tão rapidamente pode ser uma grande vantagem para aplicações epidemiológicas, como no caso da proteína spike do SARS-CoV-2. Marchand também vê potencial para o pipeline na facilitação do desenvolvimento de proteínas receptoras de antígenos quiméricos (CAR-T), que podem ser projetadas para permitir que as células imunológicas do paciente atinjam as células cancerígenas.
“Avanços adicionais nos métodos de aprendizado de máquina ajudarão a melhorar nosso método, mas nosso trabalho hoje já fornece uma estratégia para desenvolver terapias inovadoras para beneficiar os pacientes por meio do design rápido de terapias baseadas em proteínas – diretamente do computador”, concluiu o doutorando.
Acesse o artigo científico completo (em inglês).
Acesse a Notícia completa na página da Escola Politécnica Federal de Lausanne (em inglês).
Fonte: Celia Luterbacher, EPFL. Imagem: As superfícies das proteínas variam amplamente e são dinâmicas, tornando difícil prever como e onde ocorrerão os eventos de ligação. Fonte: EPFL/Ella Maru Studio.
Em suas publicações, o Canal Farma da Rede T4H tem o único objetivo de divulgação científica, tecnológica ou de informações comerciais para disseminar conhecimento. Nenhuma publicação do Canal Farma tem o objetivo de aconselhamento, diagnóstico, tratamento médico ou de substituição de qualquer profissional da área da saúde. Consulte sempre um profissional de saúde qualificado para a devida orientação, medicação ou tratamento, que seja compatível com suas necessidades específicas.
Os comentários constituem um espaço importante para a livre manifestação dos usuários, desde que cadastrados no Canal Farma e que respeitem os Termos e Condições de Uso. Portanto, cada comentário é de responsabilidade exclusiva do usuário que o assina, não representando a opinião do Canal Farma, que pode retirar, sem prévio aviso, comentários postados que não estejam de acordo com estas regras.


Por favor, faça Login para comentar