Notícia
Nova análise de DNA fornece o primeiro genoma preciso da tuberculose
Cientistas sequenciaram pela primeira vez o genoma de uma cepa de tuberculose em 1998, mas nunca tinham conseguido gerar o tipo de sequência completa e precisa que maximizaria as chances de erradicar a doença
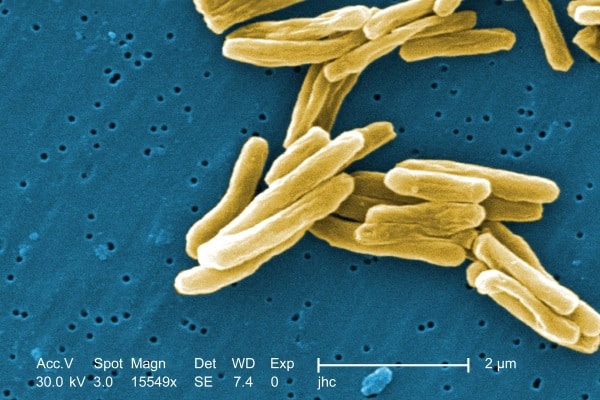
Janice Haney Carr, Public Health Image Library (PHIL)/CDC
Fonte
Universidade Rutgers
Data
terça-feira, 20 dezembro 2022 06:30
Áreas
Bacteriologia. Bioinformática. Biologia. Biomedicina. Biotecnologia. Genética. Genoma. Imunologia. Microbiologia. Saúde Pública.
Pesquisadores desenvolveram uma nova ferramenta de montagem do genoma que pode estimular o desenvolvimento de novos tratamentos para tuberculose e outras infecções bacterianas.
A nova ferramenta, que criou um mapa melhorado do genoma de uma cepa de tuberculose, deve fazer o mesmo para outras cepas e outros tipos de bactérias, de acordo com os pesquisadores, cujas descobertas foram publicadas na revista científica Nature Communications.
Mycobacterium tuberculosis, a bactéria responsável pela tuberculose, infecta cerca de um quarto da população mundial e matou 1,6 milhão de pessoas em 2021, segundo a Organização Mundial da Saúde. As intervenções médicas atuais são limitadas a uma vacina centenária que reduz o risco de infecção em 20% e quatro a seis meses de antibióticos fortes que às vezes se mostram ineficazes.
“A chave para vencer esta doença é entendê-la, e a chave para entendê-la está em seu DNA”, disse o Dr. David Alland, autor sênior do estudo e chefe da Divisão de Doenças Infecciosas da Escola Médica da Universidade Rutgers, nos Estados Unidos, e diretor do Instituto de Pesquisa em Saúde Pública da escola. “Esperamos que nosso novo pipeline forneça aos pesquisadores de todo o mundo as informações necessárias para criar tratamentos mais rápidos e eficazes e, idealmente, uma vacina totalmente eficaz”.
Os cientistas sequenciaram pela primeira vez o genoma de uma cepa de tuberculose – H37Rv – em 1998, mas nunca conseguiram gerar o tipo de sequência completa e precisa que maximizaria as chances de erradicar a doença – até agora.
O novo pipeline, chamado de Bact-Builder, combina programas comuns de montagem de genoma de código aberto em uma ferramenta nova e fácil de usar, disponível gratuitamente no GitHub.
Os cientistas hoje tipicamente sequenciam novos genomas bacterianos cortando grandes pedaços de DNA em fragmentos pequenos e rápidos de escanear e, em seguida, usam uma sequência de referência como H37Rv para alinhar adequadamente todos os pedaços de dados resultantes. No entanto, a montagem de genomas sem referência, como o Bact-Builder faz com os dados dos sequenciadores MinION, permite aos pesquisadores identificar genes presentes em cepas clínicas que podem não estar presentes na referência.
A sequência da tuberculose criada pelo Bact-Builder contém aproximadamente 6.400 mil informações (pares de bases) a mais do que a referência antiga e, mais importante, identifica genes novos e fragmentos de genes ausentes na referência antiga.
“Apenas publicar um genoma totalmente preciso para a cepa de referência H37Rv, que é usada em centenas de estudos por ano, deve ajudar significativamente a pesquisa da tuberculose”, disse o Dr. David Alland.
Ter uma maneira fácil de sequenciar todas as cepas com precisão é ainda mais importante, disse o pesquisador, “porque a comparação de cepas deve responder a muitas questões vitais, como por que algumas cepas são mais contagiosas do que outras. Por que algumas cepas causam doenças mais graves? Por que algumas cepas são mais difíceis de curar? As respostas para todas essas perguntas, que podem nos ajudar a criar melhores tratamentos e vacinas, estão no código genético, mas você precisa de uma maneira precisa de encontrá-las”.
Acesse a página do Bact-Builder no GitHub (em inglês).
Acesse o artigo científico completo (em inglês).
Acesse a notícia completa na página da Universidade Rutgers (em inglês).
Fonte: Andrew Smith, Universidade Rutgers. Imagem: Sob ampliação de 15.549x, imagem de microscopia eletrônica de varredura colorida digitalmente mostra uma série de bactérias Gram-positivas Mycobacterium tuberculosis. Fonte: Janice Haney Carr, Public Health Image Library (PHIL)/CDC.
Em suas publicações, o Canal Farma da Rede T4H tem o único objetivo de divulgação científica, tecnológica ou de informações comerciais para disseminar conhecimento. Nenhuma publicação do Canal Farma tem o objetivo de aconselhamento, diagnóstico, tratamento médico ou de substituição de qualquer profissional da área da saúde. Consulte sempre um profissional de saúde qualificado para a devida orientação, medicação ou tratamento, que seja compatível com suas necessidades específicas.
Os comentários constituem um espaço importante para a livre manifestação dos usuários, desde que cadastrados no Canal Farma e que respeitem os Termos e Condições de Uso. Portanto, cada comentário é de responsabilidade exclusiva do usuário que o assina, não representando a opinião do Canal Farma, que pode retirar, sem prévio aviso, comentários postados que não estejam de acordo com estas regras.





Por favor, faça Login para comentar